| Språk : |
|
| Encyclopedia gemenskap |Encyclopedia Svar |Submit fråga |Ordförråd Kunskap |Överför kunskap |
Fylogenetiskt träd |
    |
|
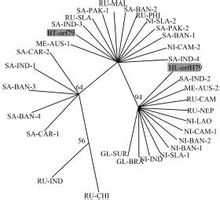
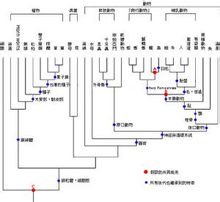
Fylogenetiskt träd (English: fylogenetiskt träd), även känd som evolutionära träd (evolutionära träd), är showen anses ha en gemensam förfader evolutionära släktskap mellan arterna trädet. Är en kladistisk metod (cladogram). I trädet representerar varje nod sin senaste gemensamma förfadern till grenen, medan längden på linjen mellan noderna motsvarar evolutionära avstånd (t.ex. uppskattningar av evolution tid).Kort introduktion Fylogenetiskt träd (English: fylogenetiskt träd eller evolutionära träd) är en indikation anses ha en gemensam förfader med varandra evolutionära släktskap mellan arter av träd, har översatts fylogenetiskt träd, fylogenetiska träd, fylogenetiska träd, fylogenetiskt träd, evolution träd, fylogenetiskt träd, systemet trädet. Det används för att ange resultaten av fylogenetiska studier, använda den för att beskriva de evolutionära sambanden mellan arter. Arter Enligt rotade och rotade att urskilja: Trädrötter kan delas in i två typer av träd och träd utan rötter. Riktningen för en rotat träd är ett träd, Innehåller en unik nod som ett träd nästan alla arter gemensam förfader. Den vanligaste metoden är att använda för att bestämma rötterna till en eller flera homologa obestritt som outgroup arter (English outgroup), utgruppen vara tillräckligt nära för att ge tillräckligt med information, men inte för nära det och trädslag blandas. För att ta bort roten rotade trädet blir orotad träd. En orotad träd när ingen annan information (outgroup) eller antaganden (t.ex. antar den maximala längden av roten grenen) inte kan bestämma sina rötter. Orotad träd är ingen riktning i vilken de två segmenten är troliga utvecklingen riktning. Gene träd och arter träd: Baserat på en enda homolog gen fylogenetiska skillnader bör anges antalet genen träd. Eftersom detta träd utgör endast en enda historia gen evolution. Istället är det i den evolutionära historien av arten. Trädslag föredras i allmänhet en analys av data från flera gener får. Till exempel uppstår en studie av utvecklingen av växter, med 100 olika gener för att bygga de trädslag, eftersom evolutionen sker i organismen befolkningsnivå, snarare än på individnivå, även om ytan inte behöver mer uppgifter, men i själva verket finns det fortfarande behövs. Gene träd och arter skillnader mellan trädet är mycket viktigt, om så bara för att bygga ett antal arter alleler som många alla kommer att grupperas tillsammans och gorillor, snarare än grupperas tillsammans med andra. Tolkning System är en gren av träddiagram (engelska cladogram). I trädet representerar varje nod sin senaste gemensamma förfadern till varje gren, och längden på linjen mellan noderna motsvarar evolutionära avstånd (t.ex. uppskattningar av evolution tid). Fylogenetiskt träd kallas ibland också dendrogram, som består av en serie av noder och grenar av. Där varje nod representerar en klassificeringsenhet motsvarar grenen nod till slutet av en gen eller organism. Med externa noder motsvarar sluta intern nod representerar en gemensam förfader. Fylogenetiskt träd struktur grundläggande informationen i ett datorprogram används ofta en kapslad uppsättning parenteser som blir Newick format. Tree algoritm Ritas med SSU rRNA fylogeni träd, de tre största grenarna (domäner) är bakterier, arkéer och eukaryoter. Matrix: granne bindningsanalys granne-anslutning (NJ) Parsimony: maximal parsimony maximal sparsamhet (MP) Maximum likelihood metoden: maximum likelihood (ML) Posteriori metod: Bayesian Konstruktion Metod Att bygga ett fylogenetiskt träd (phyligenetic träd). Fylogenetiskt träd konstruerades algoritm main Att delas in i två kategorier: oberoende element metoden (diskreta tecken metoder) och avstånd beror Frankrike (avstånd metoder). Element Metoden är en så kallad oberoende evolutionära trädtopologi bestäms av formen på varje stat att besluta om sekvensen, medan avståndet kriterium avser den form av evolutionära trädet topologier från den parvisa evolutionära avståndet beslut. Evolutionära gren längd stapel representerar evolutionära avståndet. Oberoende elementmetoden inklusive maximal parsimony (Max sparsamhet metoder) och den maximala sannolikheten metoden (maximum likelihood metoder), avstånd förlitar lag, däribland ex-matchning metod (UPGMAM) och granne sammanfogning (Neighbor-anslutning). Bygga uppgifter Används för att konstruera fylogenetiska träd uppgifter i två kategorier: funktionsanvändningsdata (närvaro av olika tillstånd av ändliga egenskap) och data avstånd (två skillnader mellan de uppgifter om alla tjugotvå åtgärd). När alla möjliga tillstånd för att avgöra likheten mellan standard, är det lätt omvandlas till funktionen data från. Bedömningsmetoder På den evolutionära trädet bedömning, främst Bootstraping Frankrike. Phylogenetic trädstruktur Construction är ett statistiskt problem, är konstruerad av det evolutionära trädet den verkliga utvärderingen av det evolutionära förhållandet eller simuleringar. Om du använder en lämplig metod, då det evolutionära trädet kommer att byggas nära den verkliga "evolutionära trädet." Simulerad evolutionära trädet behöver en matematisk metod för att utvärdera den. Olika algoritmer för olika syften. I allmänhet är det maximala parsimony metoden lämpar sig för flera sekvens uppfyller följande villkor: Jag vill jämföra skillnaderna i sekvensen av små, II för varje punkt på sekvensen har ungefär lika mutationshastighet, III utan överdriven transversion / konvertering tendens, IV av det större antalet av testsekvenser, analys med användning av maximal sannolikhet sekvens av många av ovanstående villkor inte krävs, men denna metod är mycket tidskrävande. Om analysen av sekvenserna är mer benägna att spendera några dagar till beräknat. UPGMAM (Oviktat pair gruppen metoden med aritmetiska medelvärdet) ansvara för alla punkter i den evolutionära processen har samma mutation takt, vilket är förekomsten av en molekylär klocka. Den fylogenetiska trädet algoritmen är inte särskilt exakt, relativt sett, 2013 sällan har använts. Granne sammanfogning är en ofta använd algoritm, som fylogenetiskt träd konstrueras relativt noggrann och snabb beräkning. Nackdelen är att alla platser på sekvensen behandlas lika, och att den analyserade sekvensen evolutionära avståndet inte är för stor. Dessutom måste du påpekas att för vissa specifika multipel sekvens objektet är förmodligen inte en enda existerande algoritm är mycket lämplig för det. Tillförlitlighet På avstånd metoden, är kopplingen klustringsmetod relativt enkel, är icke-vägda genomsnittliga metoden mer praktisk gruppering, när avståndet uppgifterna härrör från resultaten av analysen av flera gener, kan användningen av icke-vägda genomsnittliga metoden grupperas pålitliga fylogenetiska träd. För diskret inslag analysmetod, om sekvensdivergens i mindre utsträckning, är det en bra maximal parsimony fylogenetiskt träd byggmetod. Men i olika härstamningar större skillnad mellan graden av evolutionen, och evolutionära hastigheten konstant och trädgrenar mycket kort fall, maximal sparsamhet och inte kan verkligen göra ett fylogenetiskt träd konsekventa domar. Den fylogenetiska träd konstrueras, statistisk analys av fel som kan påverka tillförlitligheten i resultaten. Oavsett om det är ett avstånd-baserade fylogenetiska träd Rekonstruktionsmetoderna eller funktion-baserade fylogenetiskt träd metoder återuppbyggnad, kan vi inte garantera att hon kommer att få en beskrivning av den följd evolutionära historia än den sanna trädet. Ett stort antal simuleringsmetoder kan jämföra dessa landvinningar statistisk tillförlitlighet, är simuleringsresultat sammanfattas enligt följande: I allmänhet, för en datamängd, om ett sätt att sluta rätt fylogenetiska förhållandet, andra populära metoder som används att få bättre resultat. Men om de analoga dataunderlaget sekvensförändringar kraftigt, förändringstakten eller olika branscher är olika, är metoden inte särskilt tillförlitliga. Huvudregeln är att använda annan metod avstånd matris och parsimony analys av en datamängd, om vi kan producera liknande fylogenetiskt träd, då detta träd kan anses vara ganska tillförlitlig. I praktiska tillämpningar, utvärdering av tillförlitligheten i ett fylogenetiskt träd, innebär detta två problem, nämligen träd och dess komponenter (filial) nivå av förtroende är numret? Så få rätt träd är mer sannolikt än ett slumpmässigt valt rätt trädet mycket större möjlighet? Det finns många sätt att lösa dessa två problem, som kallas bootstrap metoden (bootstrapping) effektiv resampling teknik för att lösa det första problemet hade blivit den huvudsakliga metoden, medan de två enkla parametrar mellan träden är att lösa andra Ett typiskt förfarande är ett problem. |
| Användare Omdöme |
|
Inga kommentarer |